Перейти к:
GATCGGenerator: новый генератор для создания квазислучайных нуклеотидных последовательностей
https://doi.org/10.23947/2687-1653-2023-23-3-296-306
Аннотация
Введение. В последние десятилетия знания о ДНК все шире применяются для решения небиологических задач (вычисления с помощью ДНК, долговременное хранение информации). В первую очередь речь идет о случаях, когда необходимо подобрать искусственные нуклеотидные последовательности. Для их создания используются специальные программы. Однако существующие генераторы не учитывают физико-химические свойства ДНК и не позволяют получать последовательности с явно выраженной «небиологической» структурой. Фактически они генерируют последовательности, распределяя нуклеотиды случайным образом. Целью данной работы является создание генератора квазислучайных последовательностей с особой нуклеотидной структурой. Он должен учитывать некоторые физико-химические особенности нуклеотидных структур и будет задействован при хранении небиологической информации в ДНК.
Материалы и методы. Описано новое программное обеспечение GATCGGenerator для генерации квазислучайных последовательностей нуклеотидов. Оно предоставляется как SaaS (от англ. software as a service — программное обеспечение как услуга), что обеспечивает его доступность с разных устройств и платформ. Программа генерирует последовательности определенной структуры с учетом гуанинцитозинового (GC) состава и содержания динуклеотидов. Представлена работа алгоритма новой программы. Требования к сгенерированным нуклеотидным последовательностям заданы с помощью чата в «Телеграм» (Telegram), наглядно показано взаимодействие с пользователем. Определены и обобщены различия входных параметров и получаемых в результате работы программы конкретных нуклеотидных структур. Также в сопоставлении даны временные затраты генерации последовательностей при различных входных данных. Изучены короткие последовательности, различающиеся по типу, длине, GC-составу и содержанию динуклеотидов. В табличном виде показано, как в этом случае соотносятся входные и выходные параметры.
Результаты исследования. Созданное программное обеспечение сравнили с существующими генераторами нуклеотидных последовательностей. Установлено, что генерируемые последовательности отличаются по структуре от известных ДНК-последовательностей живых организмов, а значит, могут быть использованы в качестве вспомогательных или маскирующих олигонуклеотидов, пригодных для молекулярно-биологических манипуляций (например — реакции амплификации), а также для хранения в молекулах ДНК небиологической информации (изображений, текстов и т. д.). Предложенное решение дает возможность формировать специфические последовательности длиной от 20 до 5 000 нуклеотидов с заданным числом динуклеотидов и без гомополимерных участков. Более жесткие условия генерации снимают известные ограничения и позволяют создавать квазислучайные последовательности нуклеотидов по заданным входным параметрам. Кроме количества и длины последовательностей можно заранее определить GC-состав, содержание динуклеотидов и природу нуклеиновой кислоты (ДНК или РНК).
Приводятся примеры коротких последовательностей, различающихся по длине, GC-составу и содержанию динуклеотидов.
Полученные 30-нуклеотидные последовательности прошли проверку. Установлено отсутствие 100-процентной гомологии с известными ДНК-последовательностями живых организмов. Максимальное совпадение наблюдалось для сгенерированных последовательностей длиной 25 нуклеотидов (сходство около 80 %). Таким образом доказано, что GATCGGenerator может с высокой эффективностью генерировать небиологические нуклеотидные последовательности.
Обсуждение и заключение. Новый генератор позволяет создавать нуклеотидные последовательности in silico с заданным GC-составом. Решение дает возможность исключить гомополимерные фрагменты, что качественно улучшает физико-химическую стабильность последовательностей.
Ключевые слова
Для цитирования:
Кирьянова О.Ю., Гарафутдинов Р.Р., Губайдуллин И.М., Чемерис А.В. GATCGGenerator: новый генератор для создания квазислучайных нуклеотидных последовательностей. Advanced Engineering Research (Rostov-on-Don). 2023;23(3):296-306. https://doi.org/10.23947/2687-1653-2023-23-3-296-306
For citation:
Kiryanova O.Yu., Garafutdinov R.R., Gubaydullin I.M., Chemeris A.V. GATCGGenerator: New Software for Generation of Quasirandom Nucleotide Sequences. Advanced Engineering Research (Rostov-on-Don). 2023;23(3):296-306. https://doi.org/10.23947/2687-1653-2023-23-3-296-306
Введение. ДНК является уникальным биополимером, обеспечивающим хранение, передачу и воспроизведение генетической информации в живых организмах. Молекулы ДНК состоят из четырех типов нуклеотидов, содержащих азотистые основания аденин (A), гуанин (G), цитозин (C), тимин (T). Их возможные комбинации обеспечивают нуклеотидные последовательности, формирующие функциональные генетические элементы. В молекулярной биологии и генетике основные работы ведутся с нуклеотидными последовательностями живых организмов, однако возрастает потребность в создании искусственных последовательностей, особенно при решении небиологических задач (например, ДНК-вычисления [1][2], хранение в ДНК [3], криптография [4], ДНК-метчики в гидрологии [5] и др.).
Как ожидается, к концу 2040 года объем информации достигнет нескольких йоттабайт (1024), что требует ее структурирования и хранения. Оба этих процесса существенно влияют на потребление энергетических ресурсов, а также на производство устройств хранения данных и периферийных устройств (жесткие диски, твердотельные накопители). Для хранения такого количества информации требуется более 109 кг особо чистого кремния [6], которого может не хватить. Решение видится в использовании принципов ДНК для работы с масштабными объемами данных.
Нуклеотидные последовательности легко оцифровываются путем присвоения соответствующих двоичных кодов отдельным нуклеотидам [7–11] или блокам нуклеотидов [12–14], поэтому текстовые, графические или мультимедийные файлы можно преобразовывать в последовательности нуклеотидов [15–18]. Искусственные нуклеотидные последовательности можно составить вручную или сгенерировать с помощью специального программного обеспечения (генераторы ДНК) в зависимости от решаемых задач. Некоторые генераторы ДНК разрабатывались как самостоятельные приложения, другие — как часть программных пакетов, предназначенных для решения общих [19] 1, 2, 3, 4, 5 или специфических задач [20]. Как правило, генераторы ДНК разработаны на основе комбинаторных подходов и производят случайные последовательности заданной длины гуанин-цитозинового (GC) состава. Однако такие программные решения не учитывают химические свойства нуклеотидов и не позволяют получать последовательности с определенной структурой (например, без гомополимерных участков или длинных повторяющихся мотивов). Поэтому создаваемые такими генераторами последовательности не всегда можно воспроизвести в лабораторных условиях. Кроме того, такие последовательности могут быть идентичны существующим в природе фрагментам ДНК, что вносит неоднозначность при попытках закодировать информацию небиологического характера.
Цель представленной работы — создание генератора нуклеотидных последовательностей особой структуры, которые можно применять при кодировании текстовой, графической и другой информации в молекулах ДНК.
Материалы и методы. Определены критерии, которые следует иметь в виду при создании последовательностей. Учтена необходимость варьировать GC-состав, задавать определенное количество динуклеотидов, исключить гомополимерные участки в последовательностях.
Коллектив авторов разработал программу GATCGGenerator на языке Python 3.6 (Anaconda distribution)6. Для создания бота7 в «Телеграм» (Telegram) использовали Numpy 1.19 [21] и библиотеку Python GATCGGenerator. Решение предоставляется как SaaS (от англ. software as a service — программное обеспечение как услуга), что открывает возможность доступа с разных устройств и платформ.
Входные параметры: количество последовательностей, их длина, GC-состав и содержание динуклеотидов. Генератор исключает повторы длиной от двух нуклеотидов более четырех раз. Результат представлен в виде файла CSV, который содержит следующую информацию: последовательность, GC-состав и количество всех нуклеотидов.
Повторы и гомополимерные фрагменты хранятся в виде отдельного списка. Сначала случайным образом генерируется последовательность из четырех элементов (random.choice(nuc), где nuc = 'ACGT'). Затем выполняется поиск повторов. Если встречается хотя бы один элемент из списка, выполняется новая случайная генерация. Далее рассчитывается GC- и NN-состав. Если NN-состав не соответствует заданному пользователем диапазону, парный нуклеотид заменяется случайным образом и пересчитывается GC-состав. Если последовательность соответствует входным параметрам, она записывается во множество последовательностей (sequences).
Ниже представлена работа алгоритма программы.

Требования к сгенерированным нуклеотидным последовательностям задаются с помощью чата в Telegram. Пример взаимодействия с пользователем показан на рис. 1.
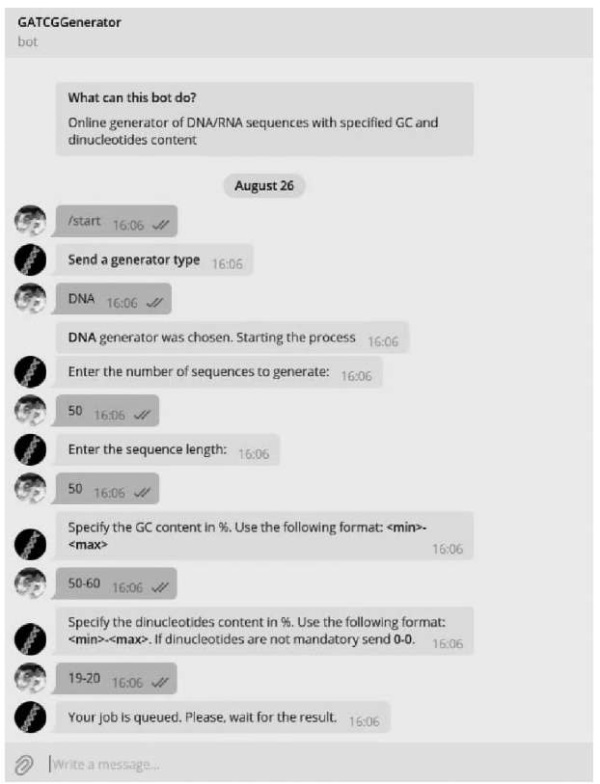
Рис. 1. Пример пользовательского чата в Telegram
В рамках представленной работы сравнивались функциональные возможности генераторов случайных последовательностей и GATCGGenerator. Определялись различия входных параметров и получаемых в результате работы программы конкретных нуклеотидных структур (таблица 1).
Таблица 1
Сравнение функциональных возможностей GATCGGenerator
с другими генераторами нуклеотидных последовательностей
|
GATCGGenerator [20] |
Nucleotide Sequence Generator8 |
DNA Sequence Tools: Random Sequence Generator9 |
Random DNA Sequence Generator10 |
Random DNA Sequence11 |
Random DNA Generator12 |
|
|
Максимальная длина (нуклеотиды) |
5 000 |
1 000 000 |
10 000 |
1 000 |
||
|
Число последовательностей |
100 |
1 |
1; 10; 50; 100 |
100 |
||
|
Ввод GC-состава (%) |
+ |
- |
+ |
- |
+ (*) |
|
|
GC- состав (%) |
интервал |
число |
- |
число |
||
|
Ввод NN-состава (%) |
- |
|||||
|
Отсутствие гомополимерных участков |
+ |
|||||
|
Тип последовательности |
ДНК/РНК |
ДНК |
ДНК/РНК/ Протеин |
ДНК |
||
|
Вывод результатов |
.CSV file |
Текст на экране |
||||
|
(*) Пользователь вводит AT-состав |
||||||
GATCGGenerator обладает более широким функционалом, дает возможность пользователю указывать количество динуклеотидов, создавать последовательности без протяженных гомополимерных участков и повторов, влияющих на успешность эксперимента. В существующих генераторах возможно только варьирование GC-состава.
Программа, созданная авторами данной научной работы, генерирует заданное количество квазислучайных последовательностей нуклеотидов, не имеющих гомологии с природной ДНК, но пригодных для молекулярно-биологических манипуляций.
Результаты исследования. GATCGGenerator позволяет генерировать специфические последовательности ДНК или РНК длиной от 20 до 5 000 нуклеотидов, содержащие заданное количество динуклеотидов и не содержащие гомополимерных участков (не более двух одинаковых нуклеотидов, расположенных рядом). Более жесткие условия генерации могут привести к длительному подбору последовательностей. В качестве примера приведем небольшой диапазон возможного содержания гуанина и цитозина и динуклеотидов (допустим, GC-состав 45–50 % и NN-состав 10–20 %). Продолжительность работы программы для различных входных данных представлена в таблице 2.
Таблица 2
Временные затраты генерации последовательностей при различных входных данных
|
Входные данные |
Время, с | |||
|
Длина |
Число |
GC, % |
NN, % |
|
|
20 |
10 |
50–60 |
20–50 |
3,45 |
|
30 |
10 |
50–60 |
20–50 |
3,91 |
|
20 |
10 |
50–60 |
40–50 |
9,74 |
|
30 |
10 |
50–60 |
40–50 |
9,53 |
|
30 |
10 |
40–50 |
20–20 |
8,80 |
|
1 000 |
100 |
45–50 |
40–50 |
11,49 |
|
2 000 |
100 |
45–50 |
10–20 |
240,25 |
|
5 000 |
100 |
50–60 |
20–50 |
11,57 |
GATCGGenerator благодаря более жестким условиям генерации последовательностей снимает ограничения известных генераторов ДНК и создает квазислучайные последовательности нуклеотидов в зависимости от заданных входных параметров. Можно указать необходимое количество последовательностей, их длину,
GC-состав и содержание динуклеотидов, а также природу нуклеиновой кислоты (ДНК или РНК). Например, созданные с помощью GATCGGenerator последовательности могут быть использованы в ДНК-стеганографии, применяемой для защиты и передачи информации путем сокрытия содержания сообщения в последовательности нуклеотидов [3].
Предлагаемое программное решение (GATCGGenerator) позволяет получать набор квазислучайных последовательностей нуклеотидов в зависимости от заданных пользователем входных параметров (тип нуклеиновой кислоты, длина последовательности, GC- и динуклеотидный состав). GATCGGenerator исключает наличие любых нуклеотидных повторов и гомополимерных участков длиннее трех элементов. Сгенерированные последовательности могут быть использованы как служебные или маскирующие (например, в ДНК-стеганографии) и подходят для любых небиологических ферментативных манипуляций. Можно сгенерировать множество искусственных нуклеотидных последовательностей и использовать их для создания универсальной олиготеки, пригодной для многократного кодирования небиологических данных и их длительного хранения.
Данные, представленные в таблице 3, обобщенно демонстрируют результаты работы программы. Для определенного типа нуклеиновой кислоты (в данном случае ДНК) показаны: содержание динуклеотидов (NN %), количество сгенерированных последовательностей, их длина (нуклеотиды — нт) и GC-состав.
Полученные 30-нуклеотидные последовательности проверили с помощью инструмента Blast от NCBI. Выявлено отсутствие 100-процентной гомологии с известными ДНК-последовательностями живых организмов. Максимальное совпадение наблюдалось для сгенерированных последовательностей длиной 25 нуклеотидов (сходство около 80 %). Это свидетельствует о способности GATCGGenerator с высокой эффективностью генерировать небиологические нуклеотидные последовательности. Можно считать, что сгенерированные таким образом последовательности не имеют абсолютного совпадения с нуклеотидными фрагментами живых организмов.
Таблица 3
Примеры коротких последовательностей,
различающихся по длине, GC-составу и содержанию динуклеотидов, %
|
Входные параметры |
Нуклеотидная последовательность, 5’→3’ |
Выходные параметры |
||||||
|
Тип |
Число |
Длина,нт |
GC, % |
NN, %* |
Длина, нт |
GC, % |
NN, %* |
|
|
ДНК |
5 |
30 |
41–50 |
20 |
CTGG**TATATCGGAATCATATCGCGCAGTGT |
30 |
46,7 |
20,0 |
|
AATCAGCTAGTAGGACGCAGTAGTGAATCA |
30 |
43,3 |
20,0 |
|||||
|
GAATGTAGTCCTAGGCACATACTACGTAGC |
30 |
46,7 |
20,0 |
|||||
|
AGTTGCACTGAAGTCTATGATCTGGCATGC |
30 |
46,7 |
20,0 |
|||||
|
GACACACTACTATGGACGTGAGGCACTTAC |
30 |
50,0 |
20,0 |
|||||
|
5 |
51–60 |
TCAGCTCAGCGCCAATCGAGCTTATAGTGC |
30 |
53,3 |
20,0 |
|||
|
GAGGCTATCGTCAAGCATAGACCGTGTGCT |
30 |
53,3 |
20,0 |
|||||
|
GACTCAGTAGCTGCTCCGGACATACAGCCT |
30 |
56,7 |
20,0 |
|||||
|
TCGCGCGTTAGACTTAGGTCTCATCGCAGC |
30 |
56,7 |
20,0 |
|||||
|
ACGCTCACAGGAGTTCGCATCGAACGATGC |
30 |
56,7 |
20,0 |
|||||
|
5 |
41–50 |
0 |
ACGACAGTGATATAGCACGACGTGCTCATA |
30 |
46,7 |
0,0 |
||
|
GACTACATCTGATAGTACACGTGCTGCACT |
30 |
46,7 |
0,0 |
|||||
|
TCTATCTCTGCTAGAGCGCTCGTCACTCTA |
30 |
50,0 |
0,0 |
|||||
|
TCTGATCTACTATAGCGATACGTGAGAGTG |
30 |
43,3 |
0,0 |
|||||
|
ACACATATATCGACGCACGCGTCGTAGTAC |
30 |
50,0 |
0,0 |
|||||
|
5 |
50 |
41–60 |
20 |
TGCATGACCATGCTTGCGGTAGACATTCAGACGCGCGAATAGTAGGACGA |
50 |
52,0 |
20,0 |
|
|
GCATACGAGTGGCATACATATTAGACTATACGGTAGTGCATATGGTGCAA |
50 |
42,0 |
20,0 |
|||||
|
CTGAGACTCCTCTCTGTGGAGCTCCTAGTACCGTCACGCGTGCTCTGAAG |
50 |
58,0 |
20,0 |
|||||
|
CTGTGTGAACATACGATGCATTCTCATCTCGGTATGGCTGAAGTGCACAT |
50 |
46,0 |
20,0 |
|||||
|
GCGCTGACGTCATGGTTCATACCAATGTAGCATGATGTGCGATAGGCACA |
50 |
50,0 |
20,0 |
|||||
|
*NN показывает долю (%) содержащихся динуклеотидов в нуклеотидной последовательности. |
||||||||
В этом случае в качестве удобного носителя информации можно задействовать специальные ДНК-олигонуклеотиды искусственного происхождения, содержащие информативную и служебную части. Недавно авторы данной работы предложили использовать NYRN-олигонуклеотиды [14], состоящие из:
- внутренней части (YR)n, кодирующей зашифрованную информацию;
- служебных (вспомогательных) частей S1 и S2, фланкирующих последовательность (YR)n (рис. 2).

Рис. 2. Структура NYRN-олигонуклеотидов:
N — вырожденные нуклеотиды;
Y — пиримидины (C или T);
R — пурины (A или G);
k, n, m — индексы, соответствующие длине части
Длина участков (n, k и m) может варьироваться, но структура служебных частей должна обеспечивать успешное протекание реакций амплификации (длина более 18 нт, 40–60 % GC-состав, отсутствие гомополимерных участков и повторов). GATCGGenerator позволяет включать динуклеотиды NN, содержащие одинаковые парные нуклеотиды (например, AA, GG, CC, TT или UU для РНК), которые могут повысить специфичность молекулярной гибридизации нуклеиновых кислот.
Обсуждение и заключение. Итак, по итогам выполненной научной работы предложено программное решение (GATCGGenerator), которое в сравнении с традиционными подходами предполагает более жесткие условия генерации последовательностей. Благодаря этой его особенности снимаются ограничения известных генераторов ДНК и формируются квазислучайные последовательности нуклеотидов в зависимости от заданных входных параметров. Исследованы полученные 30-нуклеотидные последовательности. Проверка позволила установить отсутствие 100-процентной гомологии с известными ДНК-последовательностями живых организмов. Максимально (примерно на 80 %) совпали сгенерированные последовательности длиной 25 нуклеотидов.
Отметим также, что для сокрытия информации в NYRN-олигонуклеотидах, необходимо их смешать с маскирующей ДНК. Маскирующие последовательности должны быть аналогичны последовательностям NYRN-олигонуклеотидов, чтобы при попытке считывания скрытой информации невозможно было их распознать без ключевых последовательностей. Адресату должны быть известны ключевые последовательности — праймеры к служебным участкам NYRN-олигонуклеотидов. Адресат может расшифровать переданное сообщение путем выделения информативных последовательностей нуклеотидов с помощью полимеразной цепной реакции с последующим секвенированием. Набор NYRN- и маскирующих олигонуклеотидов можно легко получить с помощью GATCGGenerator, синтезировать, а затем сохранить в виде олиготеки. Для этого достаточно определить оптимальные NYRN-олигонуклеотиды с последующим заполнением олиготеки. В дальнейшем планируется проведение лабораторных экспериментов с целью апробации предложенного метода хранения небиологической информации и проверки жизнеспособности олиготек, получаемых с помощью генератора.
1. Nucleotide Sequence Generator // nucleotide-generator.herokuapp.com : [сайт]. URL: https://nucleotide-generator.herokuapp.com/ (дата обращения: 01.12.2022).
2. DNA Sequence Tools: Random Sequence Generator // molbiotools.com : [сайт]. URL: http://www.molbiotools.com/randomsequencegenerator.html (дата обращения: 01.12.2022).
3. Random DNA Sequence Generator // faculty.ucr.edu : URL: http://www.faculty.ucr.edu/~mmaduro/random.htm (дата обращения: 02.12.2022).
4. Random DNA Sequence GenScript // genscript.com : [сайт]. URL: https://www.genscript.com/sms2/random_dna.html (дата обращения: 04.12.2022).
5. Random DNA Generator // Computer software : [сайт]. URL: http://54.235.254.95/cgi-bin/gd/gdRandDNA.cgi (дата обращения: 04.12.2022).
6. Anaconda / Anaconda Inc. // anaconda.com : [сайт]. URL: https://www.anaconda.com/ (дата обращения: 20.01.2023).
7. Python telegram bot // github.com : [сайт]. URL: https://github.com/python-telegram-bot/python-telegram-bot (дата обращения: 01.12.2022).
Список литературы
1. Малинецкий Г.Г., Митин Н.А., Науменко С.А. Нанобиология и синергетика. Проблемы и идеи. Препринты Института прикладной математики им. М.В. Келдыша РАН. 2005;29:1–26. URL: http://mi.mathnet.ru/ipmp722 (дата обращения: 01.06.2023).
2. Katz E. (ed) DNA- and RNA-Based Computing Systems, 1st ed. Weinheim: Wiley-VCH; 2021. 408 p.
3. Ceze L., Nivala J., Strauss K. Molecular Digital Data Storage Using DNA. Nature Reviews Genetics. 2019;20:456– 466. https://doi.org/10.1038/s41576-019-0125-3
4. Kaundal A.K., Verma A.K. DNA Based Cryptography: A Review. International Journal of Information and Computation Technology. 2014;4(7):693–698.
5. Aquilanti L., Clementi F., Landolfo S., Nanni T., Palpacelli S., Tazioli A. A DNA Tracer Used in Column Tests for Hydrogeology Applications. Environmental Earth Sciences. 2013;70:3143–3154. https://doi.org/10.1007/s12665-013-2379-y
6. Zhirnov V., Zadegan R.M., Sandhu G.S., Church G.M., Hughes W. Nucleic Acid Memory. Nature Materials. 2016;15:366–370. https://doi.org/10.1038/nmat4594
7. Yetisen A.K., Davis J., Coskun A.F., Church G.M., Seok Hyun Yun. Bioart. Trends in Biotechnology. 2015;33(12):724–734. https://doi.org/10.1016/j.tibtech.2015.09.011
8. Dokyun Na. DNA Steganography: Hiding Undetectable Secret Messages within the Single Nucleotide Polymorphisms of a Genome and Detecting Mutation-Induced Errors. Microbial Cell Factories. 2020;19(128):1–9. https://doi.org/10.1186/s12934-020-01387-0
9. Shuhong Jiao, Goutte R. Code for Encryption Hiding Data into Genomic DNA of Living Organisms. In: Proc. 9th International Conference on Signal Processing. Beijing: IEEE; 2008. P. 2166−2169. https://doi.org/10.1109/ICOSP.2008.4697576
10. Masanori Arita. Writing Information into DNA. In book: N. Jonoska, G. Păun, G. Rozenberg (eds). Aspects of Molecular Computing. Lecture Notes in Computer Science. Berlin, Heidelberg: Springer; 2004. P. 23–35. https://doi.org/10.1007/978-3-540-24635-0_2
11. Church G.M., Yuan Gao, Sriram Kosuri. Next-Generation Digital Information Storage in DNA. Science. 2012;337(6102):1628. https://doi.org/10.1126/science.1226355
12. K.A. Schouhamer Immink, Kui Cai. Design of Capacity-Approaching Constrained Codes for DNA Based Storage Systems. IEEE Communications Letters. 2018;22(2):224–227. https://doi.org/10.1109/LCOMM.2017.2775608
13. Nozomu Yachie, Kazuhide Sekiyama, Junichi Sugahara, Yoshiaki Ohashi, Masaru Tomita. Alignment-Based Approach for Durable Data Storage into Living Organisms. Biotechnology Progress. 2007;23(2):501–505. https://doi.org/10.1021/bp060261y
14. Garafutdinov R.R., Sakhabutdinova A.R., Slominsky P.A. Aminev F.G., Chemeris A.V. A New Digital Approach to SNP Encoding for DNA Identification. Forensic Science International. 2020;317:110520. https://doi.org/10.1016/j.forsciint.2020.110520
15. Ailenberg M., Rotstein O.D. An Improved Huffman Coding Method for Archiving Text, Images, and Music Characters in DNA. BioTechniques. 2009;47(3):747–754. https://doi.org/10.2144/000113218
16. Doricchi A., Platnich C.M., Gimpel A., Horn F., Earle M., Lanzavecchia G., et al. Emerging Approaches to DNA Data Storage Challenges and Prospects ACS Nano. 2022;16(11):17552–17571. https://doi.org/10.1021/acsnano.2c06748
17. Sakhabutdinova A.R., Mikhailenko K.I., Garafutdinov R.R., Kiryanova O.Yu., Sagitova M.A., Sagitov A.M., et al. Non-Biological Application of DNA Molecules. Biomics. 2019;11(3):344–377. https://doi.org/10.31301/2221-6197.bmcs.2019-28
18. Garafutdinov R.R., Chemeris D.A., Sakhabutdinova A.R. Chemeris A.V., Kiryanova O.Yu., Mikhaylenko C.I Encoding of Non-Biological Information for its Long-Term Storage in DNA. Biosystems. 2022;(215–216):104664. https://doi.org/10.1016/j.biosystems.2022.104664.9
19. Кирьянова О.Ю., Кирьянов И.И., Гарафутдинов Р.Р., Чемерис А.В., Губайдуллин И.М. GATCGGenerator. Свидетельство о регистрации программы для ЭВМ № RU 2021667097. 2021.
20. Borzov E.A., Marakhonov A.V., Ivanov M.V., Drozdova P.B., Baranova A.V., Skoblov M.Yu. RANDTRAN: Random Transcriptome Sequence Generator that Accounts for Partition Specific Features in Eukaryotic mRNA Datasets. Molecular Biology. 2014;48:749–756. https://doi.org/10.1134/S0026893314050021
21. Harris C.R., Millman K.J., van der Walt S.J., Gommers R., Virtanen P., Cournapeau D., et al. Array Programming with NumPy. Nature. 2020;585:357–362. https://doi.org/10.1038/s41586-020-2649-2
Об авторах
О. Ю. КирьяноваRussian Federation
Ольга Юрьевна Кирьянова, ассистент кафедры цифровых технологий и моделирования
450064, Уфа, ул. Космонавтов, 1
Р. Р. Гарафутдинов
Russian Federation
Равиль Ринатович Гарафутдинов, кандидат химических наук, заведующий лабораторией физико-
химических методов анализа биополимеров
450054, Уфа, пр. Октября, 71
И. М. Губайдуллин
Russian Federation
Ирек Марсович Губайдуллин, доктор физико-математических наук, профессор, заведующий лабораторией математической химии
450075, Уфа, пр. Октября, 141
А. В. Чемерис
Russian Federation
Алексей Викторович Чемерис, доктор биологических наук, профессор, главный научный сотрудник
450054, Уфа, пр. Октября, 71
Рецензия
Для цитирования:
Кирьянова О.Ю., Гарафутдинов Р.Р., Губайдуллин И.М., Чемерис А.В. GATCGGenerator: новый генератор для создания квазислучайных нуклеотидных последовательностей. Advanced Engineering Research (Rostov-on-Don). 2023;23(3):296-306. https://doi.org/10.23947/2687-1653-2023-23-3-296-306
For citation:
Kiryanova O.Yu., Garafutdinov R.R., Gubaydullin I.M., Chemeris A.V. GATCGGenerator: New Software for Generation of Quasirandom Nucleotide Sequences. Advanced Engineering Research (Rostov-on-Don). 2023;23(3):296-306. https://doi.org/10.23947/2687-1653-2023-23-3-296-306
JATS XML














































