Перейти к:
Компьютерная программа подбора праймеров для LAMP-амплификации
https://doi.org/10.23947/2687-1653-2024-24-1-98-108
EDN: TRWNOM
Аннотация
Введение. На сегодняшний день существует множество способов амплификации нуклеиновых кислот и у каждого способа есть ряд достоинств и недостатков. Одним из наиболее популярных способов является петлевая изотермическая амплификация (Loop-mediated isothermal AMPlification, LAMP). В отличие от термоциклических реакций, таких как ПЦР (полимеразная цепная реакция), для которых требуется смена трех температурных режимов и дорогостоящее оборудование, в LAMP вся реакция проходит при одной температуре и с максимальной на данный момент скоростью. Важным компонентом проведения LAMP-амплификации являются праймеры (обычно 20–25 нуклеотидов), которые необходимо подбирать к определенному участку нуклеотидной последовательности. Известно, что последовательность ДНК содержит четыре нуклеотида: А — аденин и Т — тимин, Г — гуанин и Ц — цитозин. Вариантов перестановок этих нуклеотидов огромное множество, и проанализировать вручную такое большое количество данных практически невозможно, поэтому возникает необходимость в использовании современных компьютерных технологий. Для дизайна ПЦР-праймеров предложено более 150 компьютерных программ, в то время как для LAMP-праймеров их менее 10, и каждая из них имеет ряд недостатков, например, по длине анализируемого участка. Поэтому целью данной работы является разработка новой отечественной компьютерной программы дизайна специфичных праймеров именно для LAMP.
Материалы и методы. В основе алгоритма поиска праймеров лежит линейный поиск подстроки в строке с учетом критериев подбора праймеров для LAMP. Программный комплекс дизайна LAMP-праймеров разработан на языке программирования Python. Для работы с различными ДНК и РНК использовалась библиотека bioPython, а для разработки интерфейса — фреймворк Qt.
Результаты исследования. Предложена модификация метода прямого перебора с использованием трафаретного подхода, учитывающего GC-состав и температуру отжига праймеров в зависимости от их структуры. Разработан комплекс программ с дружелюбным интерфейсом, учитывающий критерии дизайна праймеров: получены свидетельства о регистрации программ для ЭВМ (LAMPrimers iQ № 2022617417 от 20 апреля 2022 года, LAMPrimers iQ_loop № 2023662840 от 14 июня 2023 года). Программа есть в открытом доступе по адресу: https://github.com/Restily/LAMPrimers-iQ
Обсуждение и заключение. Разработанные программные комплексы могут использоваться для исследований и анализа в области молекулярной биологии и генетики, для создания диагностических тест-систем, обеспечивающих высокую чувствительность и достоверность обнаружения специфических ДНК и РНК. Программные комплексы могут применяться в научно-исследовательских институтах и лабораториях, занимающихся амплификацией нуклеиновых кислот. Результаты оценки подобранных наборов праймеров для реакции LAMP апробированы, и эффективность рабочих наборов с помощью программы LAMPrimers iQ доказана экспериментально на примере обнаружения генетического материала коронавируса SARS-CoV-2.
Ключевые слова
Для цитирования:
Ахметзянова Л.У. Компьютерная программа подбора праймеров для LAMP-амплификации. Advanced Engineering Research (Rostov-on-Don). 2024;24(1):98-108. https://doi.org/10.23947/2687-1653-2024-24-1-98-108. EDN: TRWNOM
For citation:
Akhmetzianov L.U. Computer Program for Primer Design for Loop-Mediated Isothermal Amplification (LAMP). Advanced Engineering Research (Rostov-on-Don). 2024;24(1):98-108. https://doi.org/10.23947/2687-1653-2024-24-1-98-108. EDN: TRWNOM
Введение. Амплификация нуклеиновых кислот является ценным молекулярным инструментом не только в фундаментальных исследованиях, но и в прикладных областях, например таких, как диагностика инфекционных заболеваний, наследственных патологий, установление родства и т. д. В настоящее время методы амплификации интенсивно развиваются, расширяются и области их применения. Самым популярным и наиболее часто используемым методом амплификации является полимеразная цепная реакция (ПЦР) [1]. ПЦР — это реакция, проходящая при трех различных температурных режимах: денатурация (95 °С), отжиг праймеров (от 50° до 60 °С), элонгация (72 °С). Для быстрой смены этих режимов необходим специальный прибор — ДНК-термоциклер [2]. При этом смены температурных режимов в амплификаторе происходят не моментально, а начинаются лишь по достижении нужной температуры, и это ведет к искусственному сдерживанию протекания реакции. Как правило, продолжительность ПЦР составляет 1–1,5 часа.
Вторым по популярности методом амплификации является петлевaя изотермическaя aмплификaция (Loop AMPlification, LАMP) [3]. Для проведения LAMP достаточно водяной бани или термостата, так как реакция проходит при одной температуре, и первые результаты можно увидеть уже через 15 минут.
Как для LAMP, так и для любого другого вида амплификации ключевым компонентом являются праймеры, которые представляют собой короткую последовательность нуклеиновой кислоты и служат отправной точкой для увеличения копий определенного участка ДНК. Именно праймеры определяют, какая последовательность ДНК будет копироваться.
Основным отличием проведения LAMP является количество праймеров. Для обычной LAMP необходимо минимум четыре праймера (два внешних, два внутренних), в то время как для обычной ПЦР достаточно двух (прямой, обратный).
Для повышения специфичности и точности реакции важно правильно подобрать праймеры. Для автоматического подбора праймеров для ПЦР разработано более 150 различных компьютерных программ, которые позволяют подбирать праймеры для любых модификаций этой реакции [4]. Однако для LAMP таких программ крайне мало, не более десяти, и всего две из них доступны онлайн. Также эти программы имеют ряд недостатков, таких как ограничения по длине анализируемой последовательности, не исключают возможности образования гомо- и гетеродимеров праймеров, повторов нуклеотидов в одном праймере. И ни одна программа не учитывает сближенное расположение праймеров в одном наборе, что, в свою очередь, снижает качество праймеров и точность результатов реакции [5].
Вследствие этого актуальной задачей является разработка новой компьютерной программы, позволяющей подбирать (моделировать) качественные наборы праймеров для LAMP-амплификации с ужесточенными условиями подбора праймеров для нуклеотидных последовательностей любой длины.
Материалы и методы. Авторы оригинальной статьи [3] предложили использовать два внешних, F3 (Forward), B3 (Backward), и два внутренних праймера, FIP (Forward Inner Primer), BIP (Backward Inner Primer). Предполагалось, что внутренние праймеры имеют удвоенную длину (FIP: F1c/F2, BIP: B1c/B2) и отжигаются на четырех участках нуклеотидной последовательности. Схематично расположение LAMP-праймеров можно увидеть на рис. 1.
Внешние праймеры необходимы только на начальном этапе. Они предназначены для того, чтобы ограничить анализируемый участок нуклеотидной последовательности и образовать одноцепочечную структуру этого участка. Пара внутренних праймеров, F1c и B1c, начинают свою работу уже на втором этапе, поскольку они отжигаются после образования новых цепей ДНК.
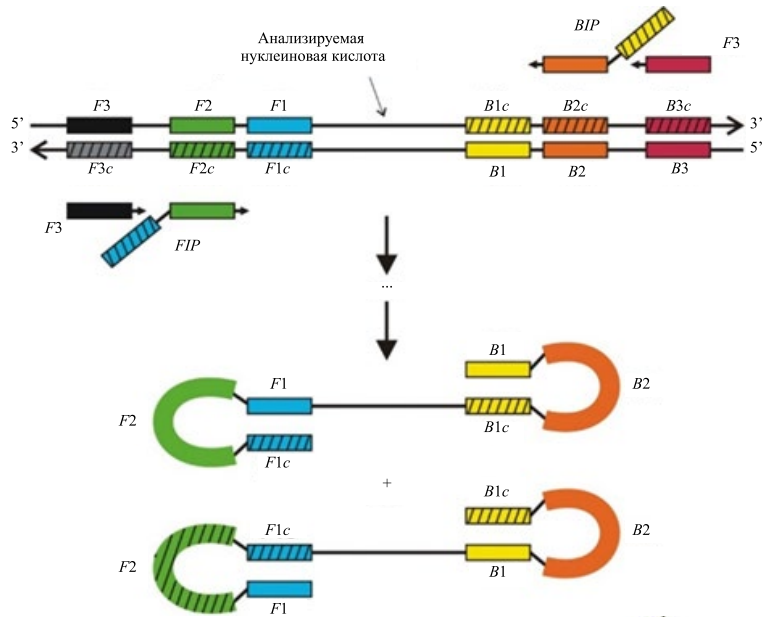
Рис. 1 Схематичное расположение мест отжига внешних и внутренних праймеров для LAMP-амплификации [6]
Позже теми же авторами был предложен модифицированный метод, предлагающий использование не четырех, а шести праймеров, отжигающихся уже на восьми участках целевой нуклеотидной последовательности [7]. Предлагалось добавить еще два петлевых праймера (Loop B, Loop F), которые должны вступить в реакцию на третьем этапе после образования гантелеобразной структуры ДНК и отжигаться между участками F1/F2 и B1/B2 соответственно. Использование дополнительных праймеров подразумевает увеличение чувствительности и достоверности реакции.
Любая реакция амплификации имеет свой порог чувствительности, и разброс данного показателя весьма велик ввиду того, что на протекание как ПЦР, так и LAMP влияет множество факторов. В некоторых работах отмечается, что LAMP по чувствительности значительно превосходит ПЦР. Например, авторы работы [8] утверждают, что LAMP чувствительнее ПЦР в 10 раз. Авторы других работ выявили, что LAMP чувствительнее ПЦР в 100 раз [9], а у некоторых этот показатель достигает 1 000 раз [10].
Помимо чувствительности, у любой реакции амплификации есть еще один не менее важный показатель — ее специфичность. И здесь к LAMP-амплификации в последнее время стали появляться вопросы [11], в том числе из-за возникновения так называемых праймерных гомо- и гетеродимеров, исключить которые в этой реакции сложнее, чем в ПЦР, ввиду большего числа используемых праймеров и их увеличенной длины [12].
Для успешного проведения амплификации необходимо правильно подобрать праймеры. При использовании метода LAMP основная сложность заключается в моделировании праймеров c учетом всех рекомендуемых условий, а именно:
- длина праймера (18–35 нуклеотидов для внешних праймеров, 30–55 нуклеотидов для внутренних);
- содержание гуанина (G) и цитозина (C) (GC-состав в пределах от 40 до 60 %);
- оптимальная температура отжига праймеров (55–65 °С);
- сближенное расположение праймеров в одном наборе: средний размер ампликона (120–220 п. н.);
- исключение образования димеров праймеров;
- исключение повторов нуклеотидов в одном праймере (не более трех).
В таблице 1 приведены краткие характеристики и возможности наиболее популярных программ дизайна LAMP-праймеров [5].
Таблица 1
Краткие характеристики популярных компьютерных программ дизайна LAMP-праймеров
| Язык разработки | Длина анализируемого участка, нуклеотиды | Подбор петлевых праймеров | Конфигурация петлевых праймеров | Графический интерфейс | Свободный доступ |
Primer Explorer (V4, V5) японской компании Eiken Chemical Co. LTD, Токио | Java | до 2 000 | + | нет | да | да |
FastPCR финской фирмы Primer Digital Ltd | Java | от 12 до 500 | – | нет | нет | нет |
университета Цзяо Тун, Китай | С, CUDA С, Perl OS Linux | не ограничено | + | + | нет | да |
американской фирмы Premier Biosoft |
| до 15 000 000 | + | только Тм | да | нет |
Python |
| – | – | – | нет доступа | |
New England Biolabs, Ипсвиче, штат Массачусетс, США | Java | от 100 до 2 000 | + | – | да | да |
LAMPrimer iQ-loop, Уфа, Россия | Python 3.10 | не ограничено | + | да | да | да |
Дизайн праймеров для LAMP-амплификации представляет собой весьма трудную задачу и требует разработки специальной компьютерной программы с должным функционалом, учетом всех рекомендуемых условий и с возможностью расширенного подбора праймеров и удобным интерфейсом.
Компьютерная программа дизайна праймеров разработана на языке программирования Python. Данный язык имеет библиотеку bioPython, которая позволяет работать с нуклеотидными последовательностями, а также фреймворк Qt для разработки интерфейса.
Результаты исследования. С учетом особенности строения нуклеотидных последовательностей и критериев подбора LAMP-праймеров была предложена модификация метода прямого перебора с использованием трафаретного подхода, учитывающего GC-состав, температуру отжига праймеров и позволяющего снизить сложность перебора.
Как известно, GC-состав праймеров должен быть в пределах от 40 до 60 %. Это один из важных критериев подбора LAMP-праймеров, который зависит от самой анализируемой последовательности, длины праймера и частично влияет на температуру отжига (Тm, °С).
В данной работе для расчета температуры отжига праймеров используется формула:
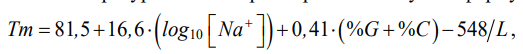 (1)
(1)
где [Na+] — молярная концентрация ионов натрия; (%G + %C) — GC-состав в анализируемой последовательности, выражается в процентах; L — длина праймера. За ее основу была взята известная зависимость:
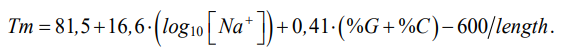 (2)1
(2)1
Формула (1) была подобрана эмпирически, расчетные значения сопоставлялись со значениями, получаемыми с помощью удобной утилиты OligoAnalyzer2, которая предоставляет качественный подбор праймеров для любых видов амплификации нуклеотидных кислот. Определив длину праймеров, GC-состав и температуру отжига, можно найти все возможные праймеры в нуклеотидной последовательности любой длины, которые будут удовлетворять заданным критериям.
Если представить праймер как подстроку, а анализируемую нуклеотидную последовательность как более длинную строку, данную задачу можно представить как перебор всех возможных вариантов (прямой поиск), но усложнив его за счет расчета GC-состава и температуры отжига праймеров.
На рис. 2 представлена полная блок-схема работы алгоритма прямого перебора, учитывающая:
- длину праймеров (п. о.);
- GC-состав, %;
- температуру отжига праймеров, Tm,°С;
- гомодимеры на обеих цепочках ДНК.
Общая сложность модифицированного алгоритма в худшем случае: O(m×n), где n — длина праймера;
m — длина нуклеотидной последовательности. Необходимо понимать, что время работы алгоритма напрямую зависит от того, насколько часто встречаются удовлетворяющие требованиям нуклеотидные фрагменты.
В таблице 2 приведены данные поиска всех возможных праймеров в нуклеотидных последовательностях различной структуры по времени.
Далее из всех найденных праймеров необходимо сформировать наборы, учитывая сближенное расстояние между праймерами, гетеродимерность в одном наборе, а также минимальную разницу температур отжига праймеров.
Схема дизайна формирования праймеров в наборы для LAMP представлена на рис. 3.
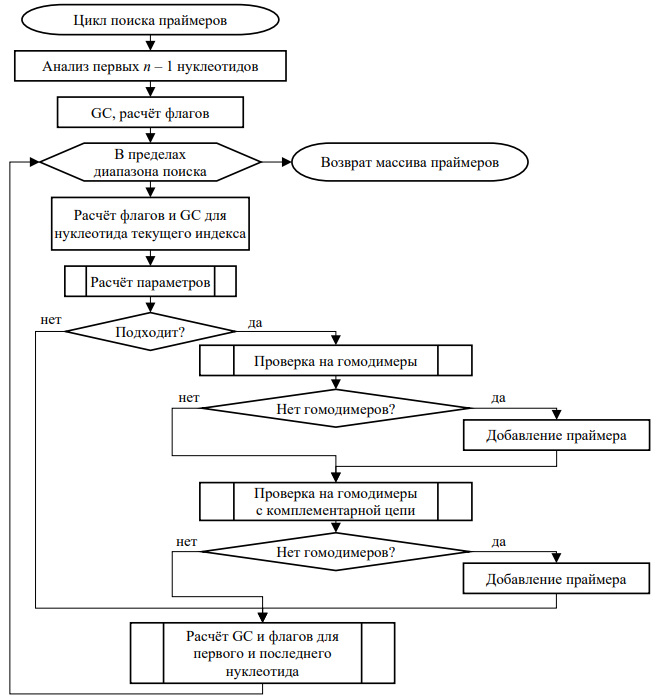
Рис. 2. Полная блок-схема работы алгоритма
Таблица 2
Время, затраченное на поиск подходящих праймеров в нуклеотидных последовательностях разной длины
Название генома | Размер нуклеотидной последовательности, п. о. | Поиск праймеров, с |
SARS-CoV-2 | 29 903 | 0,31 |
Escherichia virus T4 | 168 903 | 1,73 |
Mycoplasma | 580 076 | 5,43 |
Helicobacter pylori | 1 624 458 | 18,11 |
Escherichia coli | 4 641 652 | 71,68 (1,2 мин) |
Caenorhabditis | 100 286 401 | 1 082,53 (18 мин) |
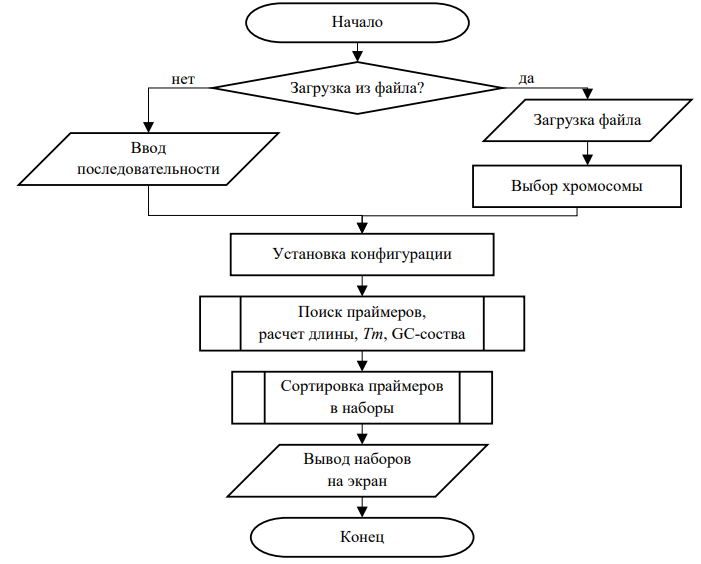
Рис. 3. Блок-схема формирования наборов праймеров для LAMP
На входе программа считывает нуклеотидную последовательность, либо загружается нужный файл, либо вставляется фрагмент через буфер обмена. Далее устанавливаются конфигурации, такие как длина праймеров, GC-состав, температура отжига праймеров и разница температур в одном наборе. Затем происходит поиск всех возможных праймеров, удовлетворяющих установленным конфигурациям, подобранные праймеры сортируются в наборы и выводятся на экран пользователю.
Работа компьютерной программы:
- Загрузка файла (простой текстовый формат, формат FASTA, GenBank) или фрагмент последовательности через буфер обмена.
- Поиск всех возможных праймеров: праймеры объединяются по следующим критериям:
- длина анализируемого участка;
- расстояния между праймерами (F3/F2 — 1–10 нуклеотидов, F2/F1с — 10–25 нуклеотидов,
F1с/B1c — 0–30 нуклеотидов); - разница температур отжига праймеров (<3);
- гетеродимерность (heterodimer).
В случае соблюдения всех вышеперечисленных условий набор считается рабочим.
- Вывод смоделированных наборов праймеров на экран пользователю и/или сохранение в файл.
Программа для LAMP-амплификации зарегистрирована в Реестре программ для ЭВМ под названием LAMPrimers iQ, № 2022617417 20 апреля 2022 года и LAMPrimers iQ-loop, № 2023662840 14 июня 2023 года. Код программы в открытом доступе3.
Разработанный программный продукт обладает дружественным и интуитивно понятным интерфейсом, что позволяет использовать его непосредственно конечным пользователям — экспериментаторам, занимающимся LAMP-амплификацией.
Обсуждение и заключение. Количество выданных наборов праймеров зависит от заданных параметров поиска. Чем строже параметры, тем меньше наборов будет найдено. В случае жестких ограничений программа может не выдать ни одного набора. Количество наборов праймеров при различных параметрах подбора для генома бактериофага лямбда [13], длина которого составляет ~48 500 нуклеотидов, представлено в таблице 3.
Для относительно коротких нуклеотидных последовательностей (до 2 000 нуклеотидов) подбор праймеров занимает менее секунды. С увеличением длины последовательности длительность поиска праймеров увеличивается экспоненциально.
Таблица 3
Количество наборов праймеров для генома бактериофага лямбда в зависимости от заданных параметров отбора
GC, % | ΔTm, °C | Максимальная длина амплифицируемого участка, п. н. | ||
300 | 230 | 160 | ||
40–60 | 5 | 213 | 119 | 3 |
2 | 198 | 184 | 3 | |
45–55 | 5 | 132 | 80 | 0 |
2 | 116 | 64 | 0 | |
50–60 | 5 | 195 | 185 | 4 |
2 | 181 | 164 | 4 | |
55–65 | 5 | 160 | 147 | 4 |
2 | 134 | 105 | 0 | |
На рис. 4 представлено влияние длины нуклеотидной последовательности на длительность подбора наборов праймеров. Приведены данные для нуклеотидной последовательности бактериофага лямбда на ноутбуке с параметрами: процессор Intel(R) Core(TM) i7-10750H CPU, 2.60GHz, 6 ядер. 16 ГБ ОЗУ. Указаны параметры мягкого подбора праймеров (40–60 % GC, ΔTm = 5, длина анализируемого участка — до 300 п. н.).
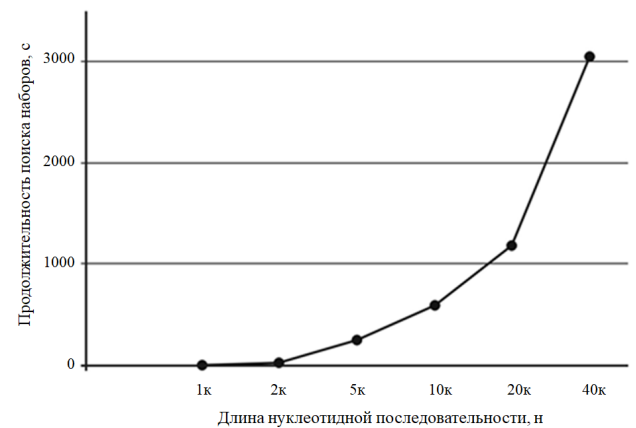
Рис. 4. Влияние длины нуклеотидной последовательности на длительность подбора наборов праймеров
Следует отметить, что продолжительность поиска праймеров зависит от мощности компьютера.
Для сравнения и определения качества смоделированных наборов праймеров был проведен ряд натурных экспериментов по обнаружению РНК коронавируса SARS-CoV-2, длина которого ~ 30 000 нуклеотидов. Для этого к одному и тому же участку нуклеотидной последовательности коронавируса были подобраны наборы LAMP-праймеров с помощью программы LAMPrimers iQ и двух популярных и доступных онлайн-утилитов от компании New England Biolabs (NEB LAMP)4 и PrimerExplorer5. Обозначения L, N и P соответствуют наборам праймеров LAMPrimers iQ, NEB LAMP Primer Design и PrimerExplorer; «+» — образцы содержали РНК коронавируса SARS-CoV-2, «–» — образцы не содержали нуклеиновых кислот. На рис. 5 приведены кривые данного эксперимента.
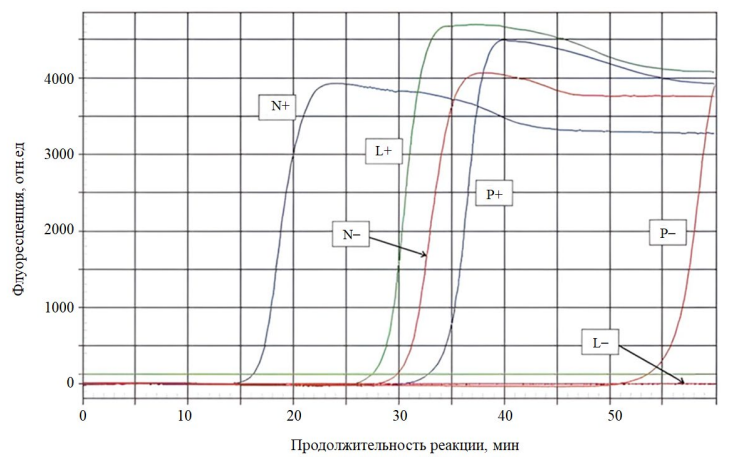
Рис. 5. График сравнительного эксперимента:
L — праймеры, полученные с помощью LAMPrimers Iq, N — NEB LAMP Primer Design;
P — PrimerExplorer, «+» — образцы содержали РНК коронавируса SARS-CoV-2;
«–» — контрольные образцы без матрицы (не содержали нуклеиновых кислот)
Праймеры, полученные с помощью PrimerExplorer, показали самый поздний подъем кривых амплификации (P+), по сравнению с NEB LAMP (N+) и LAMPrimers iQ (L+). Праймеры, полученные с помощью LAMPrimers iQ, обеспечивали более поздний подъем кривых амплификации (L+) по сравнению с (N+). Однако образцы, не содержащие РНК вируса (P–), выдали более поздний подъем по сравнению с набором (N–), в то время как (L–) не показали никаких подъёмов даже после 50 минут, тем самым обеспечивая самую высокую надежность обнаружения вирусной РНК.
Проведенные эксперименты показали более высокую точность и специфичность наборов праймеров, подобранных с помощью компьютерной программы LAMPrimers iQ, вызванную снижением скорости протекания реакции с образцами отрицательного контроля. Кривые амплификации имели более поздние подъемы или не имели их вообще, даже по истечении 50 минут продолжительности реакции.
1. Oligo Calc: Oligonucleotide Properties Calculator. URL: http://biotools.nubic.northwestern.edu/OligoCalc.html (дата обращения: 10.12.2023).
2. OligoAnalyzer™ Tool. URL: https://eu.idtdna.com/pages/tools/oligoanalyzer?returnurl=%2Fcalc%2Fanalyzer (дата обращения: 10.12.2023).
3. LAMPrimers-IQ. URL: https://github.com/Restily/LAMPrimers-iQ/blob/main/lamp/start_lamp.py (дата обращения: 10.12.2023).
4. NEB LAMP Primer Design Tool. URL: https://lamp.neb.com/#!/ (дата обращения: 24.11.2023).
5. LAMP primer designing software Primer Explorer. URL: http://primerexplorer.jp/e (дата обращения: 25.11.2023).
Список литературы
1. Гильмиярова Ф.Н., Колотьева Н.А., Гусякова О.А., Сидорова И.Ф. Полимеразная цепная реакция. История открытия. Новый этап развития. Консилиум. Лабораторная диагностика. 2017;154(4):17–21. URL: https://cyberleninka.ru/article/n/polimeraznaya-tsepnaya-reaktsiya-istoriya-otkrytiya-novyy-etap-razvitiya/viewer (дата обращения: 05.11.2023).
2. Гарафутдинов Р.Р., Баймиев А.Х., Малеев Г.В., Алексеев Я.И., Зубов В.В., Чемерис Д.А. и др. Разнообразие праймеров для ПЦР и принципы их подбора. Биомика. 2019;11(1):23–70. URL: https://biomicsj.ru/upload/iblock/ddf/bmcs19111023_1_.pdf?ysclid=lruepfy4zf297918053 (дата обращения: 05.11.2023).
3. Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, et al. Loop-Mediated Isothermal Amplification of DNA. Nucleic Acids Research. 2000;28(12):e63 https://doi.org/10.1093/nar/28.12.e63
4. Чемерис Д.А., Кирьянова О.Ю., Губайдуллин И.М., Чемерис А.В. Дизайн праймеров для полимеразной цепной реакции (краткий обзор компьютерных программ и баз данных). Биомика. 2016;8(3):215–238. URL: https://biomicsj.ru/upload/iblock/b05/3.pdf (дата обращения: 05.11.2023).
5. Ахметзянова Л.У., Давлеткулов Т.М., Гарафутдинов Р.Р., Губайдуллин И.М. Применение алгоритма Ахо-Корасик для подбора праймеров для петлевой изотермической амплификации. Математическая биология и биоинформатика. 2022;17(2):250–265. https://doi.org/10.17537/2022.17.250
6. Akhmetzianova LU, Davletkulov TM, Sakhabutdinova AR, Chemeris AV, Gubaydullin IM, Garafutdinov RR. LAMPrimers iQ: New Primer Design Software for Loop-Mediated Isothermal Amplification (LAMP). Analytical Biochemistry. 2023;684:115376. https://doi.org/10.1016/j.ab.2023.115376
7. Nagamine K, Hase T, Notomi T. Accelerated Reaction by Loop-Mediated Isothermal Amplification Using Loop Primers. Molecular and Cellular Probes. 2002;16(3):223–229. https://doi.org/10.1006/mcpr.2002.0415
8. Zhiyong Chen, Yuxue Liao, Xuemei Ke, Jie Zhou, Yixiong Chen, Lulu Gao, et al. Comparison of Reverse Transcription Loop-Mediated Isothermal Amplification, Conventional PCR and Real-Time PCR Assays for Japanese Encephalitis Virus. Molecular Biology Reports. 2011;38(6):4063–4070. https://doi.org/10.1007/s11033-010-0525-0
9. Nkere CK, Oyekanmi JO, Silva G, Bömer M, Atiri GI, Onyeka J, et al. Chromogenic Detection of Yam Mosaic Virus by Closed-Tube Reverse Transcription Loop-Mediated Isothermal Amplification (CT-RT-LAMP). Archives of Virology. 2018;163(4):1057–1061. https://doi.org/10.1007/s00705-018-3706-0
10. Wang C, Shen X, Lu J, Zhang L. Development of a Reverse Transcription-Loop-Mediated Isothermal Amplification (RT-LAMP) System for Rapid Detection of HDV Genotype 1. Letters in Applied Microbiology. 2013;56(3):229–235. https://doi.org/10.1111/lam.12039
11. Hardinge P, Murray JAH. Lack of Specificity Associated with Using Molecular Beacons in Loop Mediated Amplification Assays. BMC Biotechnology. 2019;19(1):55. https://doi.org/10.1186/s12896-019-0549-z
12. Бодулев О.Л., Сахаров И.Ю. Изотермические методы амплификации нуклеиновых кислот и их применение в биоанализе. Биохимия. 2020;85(2):174–196. https://doi.org/10.31857/S0320972520020037 Bodulev OL, Sakharov IYu. Isothermal Nucleic Acid Amplification Techniques and Their Use in Bioanalysis. Biochemistry. 2020;85(2):174–196. https://doi.org/10.31857/S0320972520020037
13. Ребольская Ю.А., Васенко Е.А., Мельник С.В., Сабиров Д.Х., Сарыгина Е.В. Бактериофаг лямбда как модельный объект в генетических исследованиях. В: Труды X Международной студенческой научной конференции «Студенческий научный форум-2018». Москва. 2018. URL: https://scienceforum.ru/-2018/article/2018002259 (дата обращения: 09.10.2023).
Об авторе
Л. У. АхметзяноваРоссия
Лиана Ульфатовна Ахметзянова, ассистент кафедры вычислительной техники и инженерной кибернетики
450062, г. Уфа, ул. Космонавтов, 1
Рецензия
Для цитирования:
Ахметзянова Л.У. Компьютерная программа подбора праймеров для LAMP-амплификации. Advanced Engineering Research (Rostov-on-Don). 2024;24(1):98-108. https://doi.org/10.23947/2687-1653-2024-24-1-98-108. EDN: TRWNOM
For citation:
Akhmetzianov L.U. Computer Program for Primer Design for Loop-Mediated Isothermal Amplification (LAMP). Advanced Engineering Research (Rostov-on-Don). 2024;24(1):98-108. https://doi.org/10.23947/2687-1653-2024-24-1-98-108. EDN: TRWNOM













































